近日,中国农业科学院李慧慧研究团队在国际期刊《Briefings in Bioinformatics》发表题为 “EXGEP: a framework for predicting genotype-by-environment interactions using ensembles of explainable machine-learning models” 的最新研究成果,提出了一套面向复杂性状预测的可解释机器学习框架 EXGEP,用于精准建模作物表型中的基因型与环境互作(G×E)效应。这是继团队今年初在《Advanced Science》上发表 AutoGS 方法后,在 G×E 建模领域取得的又一重要成果,是团队在融合人工智能与现代育种理论、构建系统化 G×E 模型体系方面的持续推进。
准确预测作物表型对于提高育种效率、缩短育种周期至关重要。然而,传统基因组预测方法往往忽视环境因素,难以对G×E带来的非线性效应精准建模,在跨环境、跨区域等泛化预测任务中精度有限,适应性不足。同时,大多数模型缺乏透明的解释机制,难以识别影响预测精度的关键影响因子。
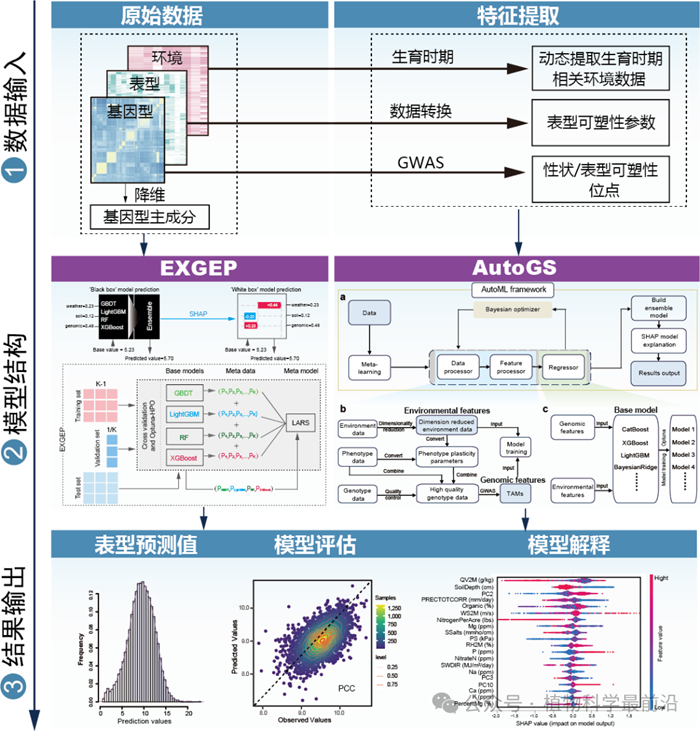
为解决这一科学问题,团队先后研发了AutoGS与EXGEP两个具有互补性的预测框架。AutoGS面向具备一定先验知识的育种场景,结合作物发育阶段划分、环境因子降维、表型可塑性建模和 GWAS 位点筛选等策略,构建精准的性状预测模型,具备较强的泛化能力,并集成超参数优化与模型融合技术。EXGEP 则更加侧重“数据驱动”的自动化建模,适用于先验信息匮乏的场景。通过自动整合基因型、气象与土壤等特征,构建高性能集成预测模型。在无需人工干预的前提下,识别影响预测性能的关键特征及其相互作用,构建特征交互网络,揭示相同特征在不同环境中的异质性贡献。AutoGS 与 EXGEP 在设计理念与应用场景上各有侧重、优势互补,系统构建了一套兼具可解释性、广泛适应性与跨物种迁移能力的G×E建模解决方案体系。该体系不仅显著提升了复杂性状在多环境条件下的预测精度,还展现出优异的泛化能力,为推动多物种、多区域和不同遗传背景下的作物遗传改良与精准育种提供了有力的人工智能工具支撑。
中国农业科学院作物科学研究所/国家南繁研究院李慧慧研究员为通讯作者,博士研究生余廷熙为第一作者。研究得到了作科所王建康研究员、 国际玉米小麦改良中心杰出科学家Jose Crossa博士、首席科学家Sarah Hearne博士、以及墨西哥科利马大学Osval A. Montesinos-López教授的支持和指导。该研究得到国家自然科学基金、中国农业科学院科技创新工程、南繁专项等项目的资助。
点击查看原文:
EXGEP: a framework for predicting genotype-by-environment interactions using ensembles of explainable machine-learning models | Briefings in Bioinformatics | Oxford Academic