2024年8月8日,Cell旗下的遗传学顶级综述期刊Trends in Genetics在线发表了中国农科院作科所/南繁院李慧慧团队联合国内外多家单位撰写的特邀综述:Artificial intelligence in plant breeding。

亮点:
1. 基因组学、表型组学和分子生物学的快速发展正在加速作物育种进入人工智能 (AI) 时代。
2. 解决管理“数据洪流”的巨大挑战对于从不同的数据集过渡到AI支持的植物育种中的集成数据基础设施至关重要。
3. 尽管AI已经彻底改变了作物育种的各个方面,如表型组学、变异检测模型、基因发掘、基因组选择和基因编辑,但迫切需要将它们协同成一种综合育种技术,以供未来作物发展。
摘要
利用尖端技术提高作物生产力是现代植物育种的关键目标。AI以其在大数据分析和模式识别方面的实力而闻名,并且正在彻底改变包括植物育种在内的众多科学领域。我们探索了AI工具在育种各个方面的更广泛潜力,包括数据收集、解锁基因库内的遗传多样性,以及弥合基因型-表型差距以促进作物育种。这将使开发适合预计未来环境的作物品种成为可能。此外,人工智能工具还有望通过提高基因编辑系统的精度和预测基因变异对植物表型的潜在影响来完善作物性状。利用人工智能支持的精准育种可以提高育种计划的效率,并有望优化基层的种植系统。这需要确定最佳的间作和轮作模式,以提高田间农业的可持续性和生产力。
在新兴技术的背景下加速遗传增益
在全球人口激增和气候变化引发的天气事件影响不断升级的情况下,提高作物产量仍然是一项艰巨的挑战。根据 Breeder 方程,遗传增益(衡量作物生产力随时间推移提高的指标)取决于选择准确性、选择强度、加性遗传变异和世代间隔的提高。在之前的工作中,我们主张利用作物基因组学、表型组学和速育方面的特定技术来加快遗传增益的速度。在这里我们假设,AI是一门跨科学学科无处不在的力量,有望加速遗传增益。
当前植物育种的格局以“数据泛滥”为特征,通过组学创新生成的数据远远超过有效的管理、归档和分析。与传统方法相比,人工智能工具可以从高通量测序和成像数据中提取更有用、更少偏见的见解。例如,基于大型语言模型 (LLM) 的 ChatGPT 是一种强大的聊天机器人,可以根据自然语言输入生成智能文本和图像。该工具在提出与植物科学相关的发人深省的问题方面发挥了重要作用,填补了植物专家在“植物科学面临的一百个重要问题”等项目中留下的空白。
除了文本处理之外,人们越来越关注在植物育种的各个方面利用LLM来加速遗传增益。组学数据类似于专门的语言输入,为训练 LLM 理解不同层次的生物过程奠定了基础。这些数据有望在预测复杂性状的价值和揭示对由不同等位基因和单倍型组成的遗传变异的生物学见解方面发挥关键作用,这些变异包括以不同的方式影响个体表型的不同等位基因和单倍型。LLMs的最新进展促使我们研究了人工智能在植物育种中的当前作用,并制定了其未来利用的路线图(图1)。具体来说,我们描绘了人工智能有可能加速遗传增益的四个关键领域:面板选择、生物大数据的生成、生物学解释和表型预测。
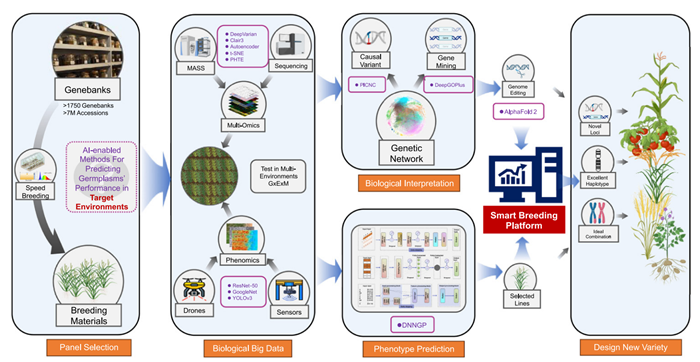
图 1. AI支持的植物育种路线图。最左边的面板描绘了来自基因库的种质资源来源,这些种质资源可以直接使用,也可以通过育种来利用组学分析生成大数据(第二个面板)。利用种质资源的大数据可以揭示基因并预测育种价值(第三个面板),从而表征基因编辑靶点和开发下一代品种的最优品系。
AI赋能的种质资源表征,生成基因组大数据
遥感和植物“组学”方面的高通量进步为科学家提供了大型多维数据集,可用于解决问题。在育种前策略、区域选择和自适应标记辅助选择中使用机器学习 (ML) 算法可以提高遗传多样性并加速具有气候适应能力的品种的开发。这些算法还可以维持和恢复导致遗传变异性的动态过程,这对于植物适应不断变化的气候是必要的。遗传增益源于作物中蕴藏的巨大生物多样性。全世界有>1750个基因库,包含>70万份种质种质,包括栽培品种、地方品种和野生近缘种,这些资源的潜力在很大程度上仍未得到开发。基因库基因组学涉及对储存的种质资源进行全基因组基因分型,为更好地了解和利用这些宝贵资源提供了一个有前途的途径。值得注意的是,已经为大量的小麦、玉米和大麦种质生成了全基因组基因分型数据,总数分别为>80000、4000和20000。这些数据集可以作为使用人工智能驱动的预测基因组学在特定环境中有针对性地选择和优化测试种质的基础。
关键的第一步包括构建一个参考基因组,作为比较物种内个体的基础资源。这使得识别、绘制等位基因突变并将其与表型多样性相关联变得更加容易,这有助于推进作物育种工作。测序技术已经产生了实质性的改进,使得能够产生更全面、更精确的参考基因组组装。10 KP计划是更广泛的地球生物基因组计划的一部分,象征着这一领域的重大进展。这个雄心勃勃的项目旨在对10000种植物物种进行测序,涵盖所有已知分类科的代表。对于其中许多物种来说,这项工作将产生它们的第一个全面的全基因组组装,这将作为后续旨在检测物种内等位基因变异的重测序工作的重要参考。大自然中固有的丰富多样性为将野生种群的有益性状整合到栽培的精英品种中提供了机会。这种整合增强了农作物和林木对不断变化的气候模式的适应能力。随着越来越多的基因组序列被编译,深入研究基因组调控和变异的潜力越来越大,并且 Ensembl 浏览器实现了对参考基因组注释的一致更新。重点将放在传播基因组数据上,以促进研究人员驱动的分析。
在不同的环境中收集如此大量的种质资源的表型数据是一项艰巨的挑战。然而,Lasky等的研究表明,利用地理参考高粱地方品种的生物气候和土壤梯度数据可以揭示与作物适应性相关的基因组特征。将基因组学与提供种质的基因型身份信息相结合,即使在没有传统表型数据的情况下,也提供了一种评估种质育种潜力的方法。增强的人工智能驱动的基因组预测模型,结合地理参考护照数据、农业气候变量和土壤梯度,可以模拟不同种质资源的性能,从而促进选择最优育种板。因此,这种方法解决了基因库种质资源利用的一个重大制约因素,即目标环境中表型信息的稀缺性。
AI驱动的数字化和表型数据收集
表型数据对于作物育种至关重要,但面临阻碍充分利用的重大障碍。长期以来,传统的植物表型分析方法因其有限的数据采集能力而被视为作物育种的瓶颈。然而,最近植物表型组学的出现代表了这种范式的根本转变。系统地研究表型的植物表型组学有望克服这些局限性。配备先进成像传感器的表型组学平台有可能彻底改变各种植物性状和环境条件的大规模表型分析(图2 A)。这些平台可以使用固定式或移动式传感器。塔架和其他固定平台通常用于监测生长阶段,因为它们简单且易于维护。为了说明这一点,安装在永久性表型分析塔上的数码相机已被用于监测水稻生长、氮含量、叶面积指数和水稻虫的存在。固定的表型分析塔易于设置和维护,但它限制了规定位置内的作物信息。由Rothamsted Research开发的用于现场表型分析的基于轨道的现场扫描器系统集成了一个传感器阵列,包括一个可见光相机、一个3D激光扫描仪、一个热红外(TIR)相机、一个叶绿素荧光传感器和一个可见的近红外高光谱相机。这种设置可以对所有生长阶段的作物冠层发育进行全面表征。此外,还引入了Crop3D高通量作物表型分析平台,该平台在可移动龙门系统内使用多个成像传感器来量化3D植物和叶片结构以及叶片温度。为了解决现场覆盖限制,传感器安装在手动推车或自行式拖拉机上。这些平台使用配备各种传感器的现象车成功捕获了大豆和小麦的冠层性状,如植株高度、归一化差值植被指数(NDVI)、温度、反射光谱和红绿蓝(RGB)图像。然而,各种环境因素(例如光强度)可能会影响开放区域的成像平台。BreedVision技术通过有效阻挡环境光并在可移动的暗室内进行成像来应对这一挑战。这种创新系统能够无损测量植物性状,包括植株高度、分蘖密度、籽粒产量、含水量和叶色。它利用一系列传感器,例如 3D 飞行时间相机、激光测距传感器、高光谱成像和 RGB 传感器。但是,根据田间拓扑、天气和土壤条件,对基于车辆的平台进行了特定限制。无人机 (UAV) 提供了一个动态平台,能够在广阔的区域快速收集数据,并生成像素密度为 ~1 毫米的高分辨率照片。高分辨率无人机摄影通过从无人机平台提供详细的冠层颜色和纹理特征以及高空间和时间分辨率,简化了表型任务。因此,高分辨率无人机摄影已在多种作物的各种表型分析任务中得到了应用,例如小麦穗识别和衰老量化。
为了在一些复杂任务(包括压力表型分析)中实现特征识别,ML 工具使用处理方法从这些大量数据中提取模式和特征。深度学习(DL)对于许多科学研究任务很有价值,因为它可以有效地识别高维数据中的复杂结构。从不同传感器获得的植物图像的广泛可变性对特定ML技术的应用提出了挑战。因此,用于特征识别的工作流程越来越依赖于深度学习工具。各种 ML 和 DL 架构,包括随机森林 (RF)、神经网络 (NN)、k 最近邻 (KNN)、偏最小二乘法 (PLS) 和支持向量机 (SVM),以及 ResNet-50、ResNetXt-101、AlexNet、DenseNet、GoogLeNet、VGG-16 和 YOLOV5 等模型,已被用于预测作物的基本性状(图 2 B)。例如,卷积神经网络(CNNs)已在RGB图像分析中应用于菊苣、小麦和油菜籽的根系分割,叶片计数,以及各种作物叶片中生物和非生物胁迫的分类和量化,以及预测单个大麦和小麦植物的穗数和产量。对于穗识别,Faster R-CCN模型使用监督学习标记训练集图像,以提高模型的准确性(图2 B)。筛选植物对不同胁迫的反应可以帮助做出选择决策,以开发具有气候适应性的品种,这是许多育种计划中的一项关键任务。CNN通过将显著性图整合到多光谱和高光谱数据分析中,对单个大豆植株的生物胁迫提供可解释的诊断。为了表征高粱对干旱胁迫的响应,研究人员使用了线性判别分析(LDA)和PLS模型。CNN通过分离模型学习的top-k特征图,生成大豆叶片中生物和非生物胁迫源的可解释分类。采用RF、NN、KNN、PLS和SVM等ML算法对水稻氮素营养指数进行估算,以提高氮素利用效率。在玉米中,使用PLS和RF模型估计地上生物量。YOLO V5-CAcT是一种创新的网络架构,已成为一种快速识别大田作物疾病的解决方案。在新型CNN(NCNN)DL平台上实施,已成为工业层面对抗作物病害的有效工具。此外,SVMs被用于3D点云分析,以估计产量和表征苹果树冠的几何形状,SVMs和高斯过程都被证明在分析TIR图像以检测菠菜干旱胁迫方面是成功的。
最近的研究表明,与单独使用单个来源相比,合并来自不同来源的数据会产生更好的结果。例如,通过深度神经网络(DNN)整合RGB、TIR和多光谱数据可提高大豆产量预测的准确性。Yoosefzadeh等还发现高度可遗传的次级特征(高光谱植被指数,HVIs)与大豆种子产量和新鲜生物量产量之间存在显著关联。它创建了集成袋装 (EB) 和 DNN 方法,以预测整个增长早期阶段的 HVI 数据。最佳HVI值与最大产量和生物量的强度帕累托进化算法2(SPEA2)有关。
使用极端学习机估计大豆氮浓度、叶面积指数、地上生物量和叶绿素含量也取得了类似的进展。为了更好地掌握理想性状和植物对环境胁迫响应背后的生物学机制,提出了将最初分开的表型组和基因组数据集的整合。这些整合工作应包括环境数据,包括气候类型,因为植物的表型受到其基因型与环境之间的相互作用的影响(G×E)。面对快速的气候变化,这种整合对于设计针对特定环境优化的作物意识形态至关重要。
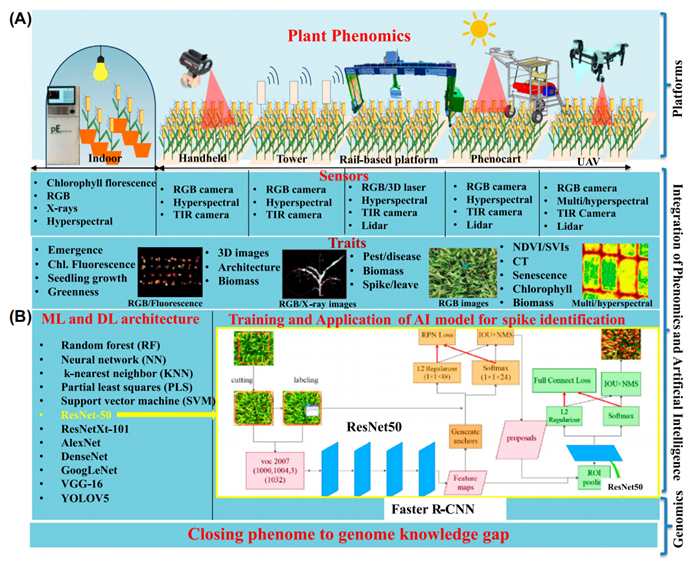
图2.植物表型分析和数据分析技术。(A) 先进的植物表型组学平台,用于表型组学数字化和大型表型组学数据集的获取。(B) 机器学习/深度学习架构,通过基于人工智能的方法预测关键植物性状,并使用基于深度学习的工作流程来识别穗数,以缩小表型组到基因组的知识差距。
AI驱动的预测来解释基因组数据
许多研究表明,人工智能在解释生化数据方面具有潜在应用,可以促进对植物胁迫生物学的理解。例如,人工智能已被有效地用于预测母代和亲本玉米植株的基因组杂交,从而有助于识别突变率较高的基因组区域。此外,基于玉米植株在胁迫下生长的DNA甲基化模式,研究人员使用人工智能方法来识别和表征基因组区域,从而区分功能基因和假基因。与此类似,Uygun等使用AI算法检查了拟南芥和玉米植物中重要基因的表达模式,以预测基因启动子和顺式调控元件。此外,通过识别生物合成基因的组织特异性变异,例如与氮利用效率、淀粉生物合成和拟南芥和水稻次生代谢物相关的变异,研究表明人工智能在破译植物代谢调控网络方面的价值。同样,Meena等证明了人工智能在生物能源管理中可以发挥的关键作用,利用人工智能通过使用各种植物物种和藻华来增加生物燃料的生产来优化生物质的产生。
测序技术日新月异,长读长测序已成为作物育种的主要方法。与短读长测序相比,该技术的优点是可以获得更多的结构变异和单倍型数据,但会给变异鉴定带来更多错误和挑战。以能够从复杂数据中提取有意义的模式为标志,提出了基于深度学习的方法,以提高短读长测序和长读长测序变异检出的准确性和效率。这些方法使用不同的策略,包括单倍型感知建模、基于图像的表示、局部重新比对和完全比对,以利用长读长中的复杂信息并减少错误的影响,表现出优于现有工具的性能,并能够在基因组的难以映射的区域中发现新的变异。一个主要的缺点是这些工具主要是在人类体细胞上训练的。表 1 中确定的变异检出需要彻底修订才能用于作物植物。显然,提高变异识别的准确性将提高基因组选择(GS)和标记辅助育种的精度,从而使育种者能够为作物改良做出明智的决策。
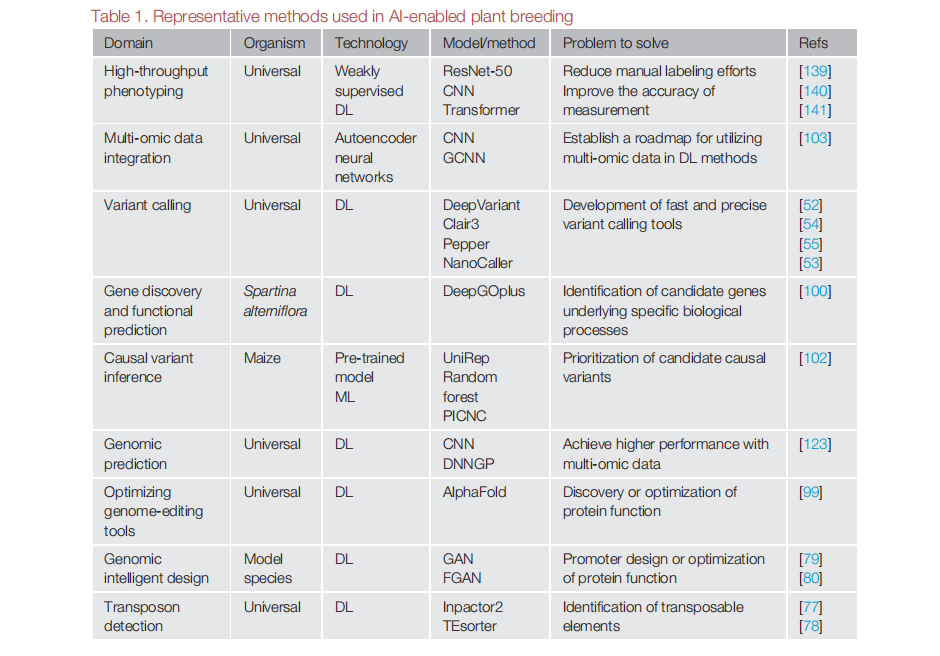
使用第二代和第三代测序技术鉴定SNP和indel仍然是一个重大挑战。人工神经网络 (ANN) 正在成为应对这一挑战的解决方案。最近,Luo等提出了一种名为Clairvoyante的CNN模型,用于预测长读长比对的SNP或插入缺失变异、合子和插入缺失长度。他们使用来自Illumina、PacBio和Oxford Nanopore测序平台的数据评估了Clairvoyante,重点是从1000基因组Project数据集中找到次要等位基因频率至少为5%的常见变异位点。
人工神经网络在变异检出中的另一个重要应用是由Poplin等人及其DeepVariant软件包证明的。DeepVariant 使用一种复杂的方法,计算每个变异位点上三种可能的等位基因组合的可能性。该工具通过分析围绕假定变体的读长图片与真实基因型检出之间的统计相关性,学习区分纯合或杂合等位基因以及变异中的参考等位基因和纯合等位基因。此外,ML算法超越了长读长测序实践中的变异检测,正如它们在群体遗传学研究中的应用所证明的那样。监督 ML 方法已成功检查了目标基因组内的重组率。例如,Schrider 和 Kern 使用射频分类器使用 DNA 基序来区分黑腹果蝇的重组率水平。基因组学和机器学习的结合,以产生预测农业疾病的创新方法,并在没有编码专业知识的情况下简化生物信息学管道,这一点非常重要。该领域值得注意的技术提供商包括 Trace Genomics 和 Sequentia Biotech。Trace Genomics 在土壤健康应用中具有特色,其中获得专利的 ML 算法用于确定影响作物性能的关键因素。相比之下,Sequentia Biotech提供AI RNA-seq(AIR)等解决方案,以简化转录组研究中的数据生成和解释程序。机器学习的未来可能需要同时处理多个物种。深度学习方法可用于从模式植物到目标作物的知识转移或比较基因组学研究等目标。
多组学大数据在植物育种中的整合
在生化水平上,“组学”涵盖了多种分子数据源,如基因组学、表观基因组学、蛋白质-DNA相互作用、转录组学、蛋白质组学和代谢组学。在过去的十年中,已经产生了大量的组学数据,使网络上充斥着转录组学、基因组学、蛋白质组学和代谢组学信息。近年来,由于高通量亚分子生物学实验在基因组学、转录组学、蛋白质组学和代谢组学等各个领域产生的大量数据集,生物学已经发展成为一门信息密集型科学。在生物信息学中,基因组、转录组、蛋白质组和代谢组水平的成分清单逐渐变得全面,对研究人员来说具有不可估量的价值。ML 经常用于评估庞大的数据集,因为它们的大小、复杂性和对组合解释的要求。即使在每种组学技术中,除了用于全基因组基因表达的RNA-seq、染色质免疫沉淀与深度测序(ChIP-seq)、DNA亲和纯化测序(DAP-seq)以及转座酶可及染色质测序(ATAC-seq)的几种分析技术也已经成熟。从单一遗传元件到调控网络,获得越来越详细的系统生物学信息,为我们提供了前所未有的机会来捕捉表型变异背后的真实生物物质。然而,为了加快作物育种速度,正确整合各种数据层,将它们与环境中的胁迫反应连接起来,并对整个系统进行精确建模仍然具有挑战性。
机器学习已证明有用的一个领域是识别各种类型的基因组区域。例如,在玉米中,活基因和死假基因可以通过根据DNA甲基化特征训练的ML模型进行分类。基于ML的方法也被开发出来,用于预测沿染色体的交叉概率。此外,ML开始在群体遗传学中找到应用,尽管主要在人类中。ML在植物中的一个应用是预测自然选择保留的突变的近乎完全固定。这些实例凸显了ML如何超越其在基因和基因组注释领域的常规作用,可用于进一步探索基因组功能,补充传统的比较基因组学方法。
AI驱动的基因型-表型差距桥梁
人们越来越关注利用人工智能对作物性状和遗传研究进行精确、无损估计的兴趣。开发具有高产潜力且对气候变化具有抵抗力的品种,在很大程度上依赖于通过鉴定新的等位基因和作物育种中的基因组辅助选择进行的遗传改良。一些研究已经成功地将基于人工智能的方法应用于高光谱和RGB数据集,以预测小麦的早期产量并进行定量基因组分析,并在小麦中鉴定出新的等位基因。Lei等使用定量遗传分析与地面相机的RGB图像实现了自动尖峰计数系统,并通过增加训练数据和注释提高了Faster R-CNN模型的准确性和泛化性(图2 B)。准确的疾病筛查对于开发抗病品种至关重要,并且已通过使用基于DL的大豆猝死综合征(SDS)检测的全基因组关联研究(GWAS)分析来解决,并揭示了候选SDS基因附近的显着SNP。此外,研究还利用多模态深度学习进行产量预测,表明表型-基因型多实例学习(PheGeMIL)、留一环境学习(LOEO)、支持向量回归(SVR)和梯度提升机(GBM)等方法利用表型观察来增强小麦产量的基因组预测。总之,整合表型组学、基因组学和人工智能提供了一种有前途的方法,可以监测作物生产力,评估对非生物和生物胁迫源的反应,以及识别新的基因和数量性状位点(QTLs)。这种综合方法可以为加快作物育种工作以开发具有气候适应能力的作物铺平道路。
“遗传力缺失”是指所有遗传变异都不能完全解释复杂性状的遗传力的情况,是遗传研究的主要瓶颈。在这种情况下,整合转座子和表观遗传突变(如DNA甲基化)可以提供更全面的信息来解释表型的遗传基础。在利用DL鉴定和分类植物转座子方面取得了巨大进展,包括Inpactor2 和TEsorter。除了利用人工智能进行基因鉴定以弥合基因组-表型组差距外,人工智能在鉴定基因的功能基因组学方面也有很广阔的应用,可以构建基因组智能设计。例如,生成对抗网络(GAN)用于通过合成启动子设计在大肠杆菌中优化和增强基因组功能。该模型由从天然启动子收集的序列特征指导,包括不同位置的核苷酸之间的相互作用,并在计算机模拟中设计出新型合成启动子。此外,反馈GAN(FBGAN)引入了一种新的反馈回路组织,通过生成编码可变长度蛋白质的合成DNA序列并使用外部功能分析器对其进行优化,从而优化蛋白质功能。仍然需要进一步的工作,以将尖端的合成方法应用于植物生物学和作物改良。
使用AI的功能基因组学和基因挖掘
已经出现了几种ML方法,以优先考虑与农艺性状相关的基因,例如利用基因功能,探索蛋白质相互作用,以及结合基因注释和序列变异。进一步探索的一个有趣途径在于将进化洞察整合到 ML 模型中。例如,最近的研究表明,利用来自注释良好的物种的知识来推断特征较差的物种的基因功能具有预测能力,特别是在预测专门的代谢基因方面。许多研究的区别在于它们双重关注优化预测性能和揭示数据背后的生物学意义特征。例如,Lin等发现转录因子在优先考虑与拟南芥和水稻特定性状相关的基因方面起着关键作用。同样,Demirci等发现,特定的DNA形状特征预测了各种植物物种(如拟南芥、番茄、玉米和水稻)的交叉发生,突出了共性和物种特异性的细微差别。一些研究利用各种机器学习模型来识别负责非生物胁迫的基因。从 ML 模型中收集的这些见解为生成可测试的假设铺平了道路,例如识别基因组区域、候选基因或蛋白质残基以进行进一步的实验验证。ML在单细胞RNA测序中发挥关键作用的一个令人兴奋的前沿,使人们能够探索复杂、异质组织中的发育过程和对环境刺激的反应。由此产生的数据集是全面的,包含来自数千个细胞和数万个基因的数据。检查此类数据通常包括无监督 ML 技术的应用。与许多预测特定“标签”的研究不同,无监督机器学习方法旨在识别有助于组织和解释缺少预定义标签的数据的模式。这些方法的例子包括聚类和流形学习方法,它们试图以类似于主成分分析的非线性方式揭示数据中的底层结构。尽管代谢组学由于几种成分的未知特性而面临挑战,但 ML 提供了整合和分析代谢组学数据的解决方案。ML方法能够预测代谢途径,例如针对西红柿的研究。正如McLoughlin等人所说明的那样,ML有望在整合转录组学、蛋白质组学和代谢组学数据的多组学分析中做出重大贡献。他们使用广泛的多组学分析来检查在充满氮和饥饿条件下培养的玉米自噬突变体,以确定细胞/组织如何依赖于自噬。即使在没有胁迫的情况下,缺乏必需自噬成分ATG12的植物在叶片代谢组中也表现出广泛的异常,重点是脂质转换产物和次生代谢产物。转录组和/或蛋白质组的重大修饰支持了这些变化。通过比较 mRNA 和蛋白质的丰度,可以确定自噬清除的特异性蛋白质靶标,以及蛋白质复合物和细胞器,并确定受这种分解代谢调节的许多过程。mlDNA R包是ML-based differential network analysis的缩写,它使用RF算法来精确定位拟南芥中与盐胁迫相关的基因。另一种创新策略涉及合并多个基因调控网络(GRNs)以及优先级算法,这导致了OsbHLH148的鉴定和验证,OsbHLH148是水稻中与干旱相关的重要转录因子。与依赖GRNs的基因发现相比,GWAS提供了一种相对直接的方法来检测与性状和自然变异相关的基因,这些变异是分子育种的自然变异。然而,由于连锁不平衡(LD),通过GWAS鉴定的QTL可能包含许多基因,这带来了挑战。选择候选基因进行验证仍然是生物学研究中的一大难点。为了应对这一挑战,已经开发了各种基于ML的靶向GWAS鉴定的QTLs的方法,用于基因优先级和因果突变的阐明。这些ML方法包括惩罚回归、梯度提升机(GBMs)、贝叶斯方法和深度学习。基于ML的基因优先级依赖于从已知基因和表型变异背后的因果变异中编译特征。以QTG-Finder为例,这是一个配备了各种ML方法的GWAS结果解释器,它能够利用一个由28个属性组成的特征集,这些属性来自拟南芥的基因组数据,包括DNA多态性、功能注释、协功能网络和进化守恒。增强版本QTG-Finder2整合了来自多个模型植物的直系同源信息,以丰富全面基因发现的特征集。QTG-Finder2有效地优先考虑通过GWAS在非模式植物物种中鉴定的基因,从而解决了因果基因信息稀缺的挑战。然而,有限的先验知识仍然是基于机器学习的植物基因发现方法的重大挑战。这个问题在未来可以通过半监督学习策略来克服,例如正无标签(PU)学习。最近的研究强调了机器学习和深度学习技术在解决特定生物学挑战方面的关键作用,特别是在鉴定植物耐盐基因方面。开发并部署了基于LLM的基因功能预测模型,以识别盐胁迫下差异表达的基因,并揭示关键通路。Yang等还利用基于DL的基因注释工具DeepGOPlus检测了与耐盐性相关的高亲和力K + 转运蛋白(HKTs),揭示了16个具有不同表达模式和离子转运偏好的互花米草(Sa)HKT基因。除了使用蛋白质序列作为输入外,Gao等还将ML与基因的协同进化信息相结合,以精确定位盐胁迫相关基因,他们的研究强调了与离子转运和解毒途径相关的基因的富集。这些发现凸显了计算方法在揭示互花米草耐盐机制的分子复杂性方面不可或缺的作用,并为植物功能基因组学提供了宝贵的见解。这些工作证明了DL和ML使用各种数据类型来研究植物非生物胁迫的关键基因的灵活性。
从组学数据中寻找优良等位基因和因果变异
在揭示生物系统中的复杂关系方面,从多组学数据构建遗传网络通常比单组学应用更有效。尽管如此,组学数据的超高维数往往会引发“维数诅咒”,对单组学数据和集成多组学数据分析构成重大挑战。因此,降维始终是多组学数据分析的第一步。Kang等确定了多组学数据集成的路线图,包括使用自动编码器对数据进行去噪和使用深度学习方法(如CNN)提取特征。尽管该路线图是针对生物医学数据提出的,但它为在植物研究中利用大型数据集提供了宝贵的试金石。凭借强大的数据集成方法,多组学分析通常可以提供比单组学分析更准确的预测。这可能是因为这些方法包括各种生物数据类型之间的复杂相互作用和依赖性,从而提供了对生物系统的更全面和整体的理解。
植物基因的功能研究,特别是那些具有农艺性状基础的基因,为通过基因改造和基因组编辑育种作物提供了重要信息。从多组学数据中表征基因功能是发现与特定过程有关的基因的基础。利用深度学习的力量,Yang等成功地利用基于深度学习的模型检查了多组学数据,并在非模型盐生植物中鉴定了一系列耐盐相关基因,并验证了其功能。深度学习也被应用于直接表征致病表型变异,这是一个具有挑战性但有价值的目标。例如,基于ML的方法PICNC(通过校准的核苷酸保守预测突变影响)可以推断由玉米突变引起的功能改变。它整合了通过UniRep获得的突变重要性信息,UniRep是一种基于长短期记忆(LSTM)的突变/蛋白质结构评估方法。基础人工智能工具可用于创建更先进的生物学解释工具。
AI驱动的基因组选择预测进行植物育种应用
标记辅助选择 (MAS) 和 GS 是两种主要的基于标记的育种技术,用于表征具有理想特性的植物。MAS在检测对复杂性状影响相对较小的基因方面面临局限性。当与这些性状相关的标记仅捕获一小部分遗传变异时,与表型选择相比,MAS可能表现不佳。
为了解决模型之间预测准确性差异的局限性,主要方法涉及使用不同的统计模型进行重复试验,以确定目标表型性状的最佳情景。GS根据对先验信息和参数设置的利用,大致分为参数方法和非参数方法。参数化方法包括正则化线性回归(RLR)模型,如岭回归(RR)和最小绝对收缩和选择算子(LASSO),它们解决了简单线性模型中固有的过度参数化问题。基于ML的统计模型,包括SVM, ANN和RF已经在植物育种中得到了应用。由于作物、栽培品种、栖息地、种群和标记各不相同,因此找到最佳统计方法存在障碍。因此,在使用 GS 时,育种者需要比较和选择适合每种情况的实用统计方法。
ML 利用统计方法使系统无需显式编程即可从数据中学习。ML 使用样本数据集生成模型来探索算法,这些算法可以从可访问的数据中学习并对看不见的数据进行预测。基于ML的方法比传统的GS具有更高的预测准确性。与传统的统计模型不同,ML 提供了灵活性,允许输入数据和结果之间的复杂关系。随着基因组数据规模的扩大和复杂性的出现,信息和预测模型的开发变得具有挑战性。因此,ML的使用正在增加,因为它提供了一种重要的替代方案,因为它在处理这些复杂性方面具有灵活性和有用性,通过修改参数模型无法包含的未识别结构的模糊模式。
传统的统计方法难以检验植物数量性状的遗传基础,特别是在涉及多效性基因、上位性和基因-环境(G x E)相互作用的复杂情景中。挑战在于识别所有标记效应,产生“大 P,小 N”问题,以及可能的过度参数化。机器学习方法提供了一种解决方案,通过利用重复的经验来提高预测的准确性。ML 算法分为有监督学习方法和无监督学习方法:监督学习旨在根据输入数据预测目标值,而无监督学习则揭示输入变量之间的分组和关联,而没有输出变量。基于ML的GS方法主要由监督学习模型组成。
SVM 是一种典型的基于 ML 的模型,在分类和回归任务中都具有优势。SVM 的与众不同之处在于它专注于在复杂多样的数据集中检测微妙的模式。SVM 使用各种特征向量开发决策边界,以产生准确的预测。这种类型的方法通过利用各种核函数来增强表型和基因型之间的非线性形成。近年来,人工神经网络方法在GS的应用中显示出了其潜力。人工神经网络可以识别数据中的模式并为复杂函数生成预测,从而充当通用逼近器。在 GS 中,这些功能可准确检测基因组标记中的上位或显性等因素。此外,它们不依赖于关于表型分布的假设,并且在GS中使用人工神经网络可以有效地估计复杂相互作用的影响。
一些研究已经使用深度学习来分析GS。Montesinos-López等使用密集耦合网络架构比较了基因组最佳线性无偏预测(gBLUP)和深度学习模型。本研究评估了 9 个已发表的基因组数据集(6 个小麦数据集和 3 个玉米数据集)。当忽略 G × E 交互时,DL 在 9 个数据集中的 6 个数据集中表现出更好的预测准确性。另一项研究发现,SVM和多层感知器(MLP)的计算效率优于其他方法。目前关于基于深度学习的GS方法的文献在比较预测准确性与传统统计方法方面很少。因此,有必要进行进一步的研究来弥合这一差距。DL通过将三个或多个人工神经网络集成到DNN结构中来构建人工神经网络。GS中流行的深度学习架构是MLP、CNN和递归神经网络(RNN)。MLP 通常是受监督的,至少集成了一个隐藏层,由于其在预测任务中的简单性和有效性,非常适合各种应用。尽管MLP具有多功能性,但在训练过程中可能会过度拟合,当应用于真实世界的数据集时,可能会降低准确性。
CNN 主要用于与计算机视觉相关的任务,这些任务将图像或视频数据作为输入。CNN 的一个关键方面是它们通过减小输入大小和参数共享来实现效率。这种优化限制了需要估计的参数数量,从而提高了计算效率。典型的 CNN 架构由三个主要操作组成:卷积、非线性变换和池化。这些操作在不影响相关信息的情况下减小了输入大小,从而通过参数减少促进了快速训练。
RNN并不是严格地在单一方向上传播:它们包含反馈环路,使信号能够通过突触连接向前和向后传播。因此,训练RNN需要大量的计算资源。DNN基因组预测(DNNGP)是一种基于深度学习的基础基因组选择方法,可以整合多组学数据来预测植物表型。该方法集成了精心设计的算法结构,以限制过拟合并提高收敛速度。它在预测准确性方面明显优于传统方法,尤其是在处理大量人口时。这种基于人工智能的工具将逐渐取代传统的植物育种方法,特别是在生物数据量呈指数级增长的背景下。利用基因功能、表达和相互作用的先验知识有助于指导基因组预测模型。这将有助于降低数据的维度和复杂性,并提高预测的生物学可解释性和可靠性。人工智能方法还可以根据可用的生物学信息(包括基因本体、转录组学和GWAS数据),使用各种策略将先验知识整合到基因组预测模型中,包括定义核函数、划分基因组变异或设计网络架构。
尽管目前只有有限数量的GS程序使用深度学习,但它越来越被认为是一种有前途的遗传预测方法。首先,深度学习模型无需预处理即可高效处理原始图像数据。其次,深度学习在没有额外预测项的情况下捕获遗传多样性,从而能够表示非累加效应和复杂的遗传关系,这对于全面的遗传评估是必要的。第三,像CNNs这样的DL结构在相邻的SNPs中获得了连锁不平衡。最后,特定的深度学习结构(如CNN)共享参数,从而减少了需要估计的参数数量。但是,在 GS 中使用 DL 伴随着一些注意事项。与典型的统计模型相比,深度学习更容易受到过拟合的影响,但可以使用贝叶斯方法来缓解这种情况。此外,由于需要选择各种超参数和涉及的调整过程,实现和优化深度学习模型需要丰富的经验。为了在GS中有效地利用深度学习,需要进一步的迭代和协作检查,以及获取更广泛的数据集。这些数据集应包括表型信息和各种组学、气候和育种者经验数据。此外,优化深度学习模型拓扑结构对于设计高效的GS框架是必要的。
AI在基因编辑中的应用
传统的育种方法,如诱变、杂交和基因工程/转基因育种,为提高作物产量和质量做出了重要贡献。然而,它们存在育种周期长、随机性高、精度低、基因功能缺失不完全、筛选过程费力等缺点。基因组测序技术的兴起为精确高效的分子育种开辟了途径,并得到了育种者的青睐。值得注意的是,CRISPR/Cas9技术的改进彻底改变了育种工作,并显著推动了作物质量提高的研究。
基因编辑系统的发展加速了分子生物学和育种学的进步。定点核酸酶 (SDN) 分为五类——归巢核酸内切酶 (HE)、巨核酸酶 (MN)、锌指核酸酶 (ZFN)、转录激活因子样效应核酸酶 (TALEN) 和 CRISPR/Cas9——在基因组编辑技术中发挥着关键作用。HEs和MNs是罕见的核酸内切酶,可识别大的DNA序列,在识别其靶位点方面构成挑战。ZFNs是第一代基因组编辑核酸酶,利用由锌离子调节的小锌指蛋白基序,这些基序以序列特异性方式与DNA结合。与 HE 和 MN 不同,多个 ZFN 可以组装成复合物,从而增强 DNA 结合特异性。同样,TALENs是通过将TALE模块融合到FokI DNA切割结构域来开发的,从而产生有效的可编程核酸酶。
AI 已被用于表征结构信息和优化蛋白质功能。AlphaFold2是一种高度精确的蛋白质结构预测工具,是人工智能在生物学中的一个显著应用,现在作为生物学研究的通用“基础设施”运行。另一项突破性技术,基因组编辑,为作物改良打开了新的大门。人工智能辅助的基因组编辑和合成生物学可能允许通过基因改造生产出理想的植物。然而,通过基因组编辑生产育种材料可能需要不断优化且效率更高的工具。Huang等利用AlphaFold2预测的蛋白质结构数据揭示了新的脱氨酶功能簇,并利用这些信息开发了更有效的碱基编辑器。最近,第一个从头生成的基因编辑器OpenCRISPR-1是由在>100万个CRISPR操纵子上训练的LLM设计的,开启了直接使用LLMs设计蛋白质的新篇章。
此外,设计具有紧凑结构的基因组编辑器对于提高基因操作的精度和效率至关重要。人工智能技术可能会彻底改变紧凑而全面的基因组编辑工具的设计。通过蛋白质结构预测方法,直接重新设计构成关键农艺性状的蛋白质更为简单。然而,优化这些蛋白质需要大量的特定训练,而确保重新设计的蛋白质在体内发挥足够的作用需要多学科合作和全面的知识图谱。然而,蛋白质结构预测工具可以帮助改进作物设计,可以合理推断,人工智能驱动的蛋白质工程将有助于未来的作物改良。
结语
人工智能技术可以彻底改变公共部门的植物育种计划(框注1),例如国际农业研究磋商小组(One CGIAR)计划下的计划。数据驱动的分散育种程序,如这些,可以比传统的GS更好地预测作物性能。人工智能支持的育种平台通过部署先进的计算和分析算法来加速育种过程。人工智能在基因发现和等位基因挖掘方面的前景是明确的;然而,人工智能的真正前景是协助未来作物品种的生物学设计,这些作物品种非常适合预测的环境。进一步的挑战将是:(i)模拟和表征预测环境的基因库的多样性,以便在没有连锁拖累的情况下将新性状引入品种,(ii)构建能力和训练有素的人力资源,以有效利用计算能力进行人工智能驱动的预测育种,(iii)为参与设计未来作物品种的育种团队提供多学科人工智能培训(框注2), (iv)建立统一的植物育种网络基础设施,而不是数据孤岛(见未决问题)。人工智能的应用在预测最佳种植模式和系统方面也显示出巨大的前景,通过整合从物理传感器、无人机平台和物联网(IoT)设备获得的大数据,这些数据在不同基因型×环境×管理实践下。这将补充基于人工智能的基因创新,以获得所需的遗传增益率,以应对未来十年的粮食和营养挑战。
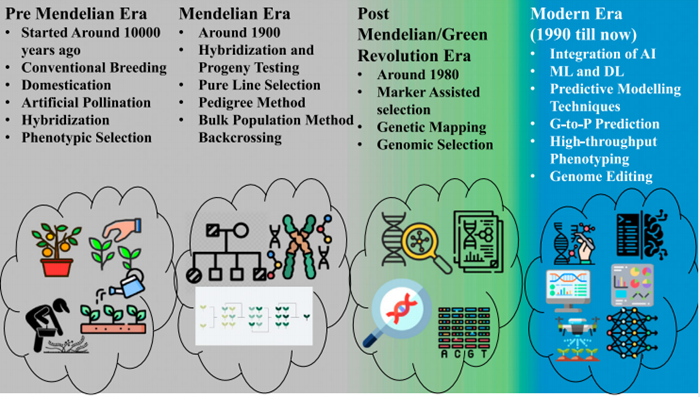
框注1. 四个植物育种时代以及每个时代的关键育种策略。
突出问题
1. 如何将人工智能实际整合到育种计划中,使育种者和农民能够利用先进技术进行作物改良?
2. 如何对人工智能技术进行定制和优化,以提取有意义的见解并推动植物表型组学研究的进步,尤其是在处理有限或较小的数据集时,从而为更精确和有针对性的作物改良策略铺平道路?
3. 人工智能驱动的图像分析和计算机视觉技术如何帮助识别植物中微妙的表型性状,最终帮助育种者选择复杂的性状,如耐旱性或抗病性?
4. 人工智能在利用不断增长的多组学数据来促进发现新的遗传资源和提高快速和精确的GS方面有什么潜力?
5. 根据一种环境或作物物种的数据训练的人工智能模型对其他环境或物种的推广效果如何?可以使用哪些策略来提高人工智能模型在不同遗传背景和环境条件下的可转移性和稳健性?
6. 植物育种课程的设计在多大程度上使未来的植物育种者能够充分利用新的人工智能技术的潜力?
7. 我们如何通过投入资源,使人工智能技术在公共部门育种计划中易于获取和使用?
8. 人工智能驱动的植物育种产品何时上市?
原文链接:
https://www.sciencedirect.com/science/article/pii/S0168952524001677
地址:海南省三亚市崖州区金稻路5号 邮编:572024 邮箱:nanfanyuan@caas.cn 电话:0898-38288010
Copyright © 中国农业科学院 京ICP备10039560号-5 京公网安备11940846021-00001号
技术支持:中国农业科学院农业信息研究所

